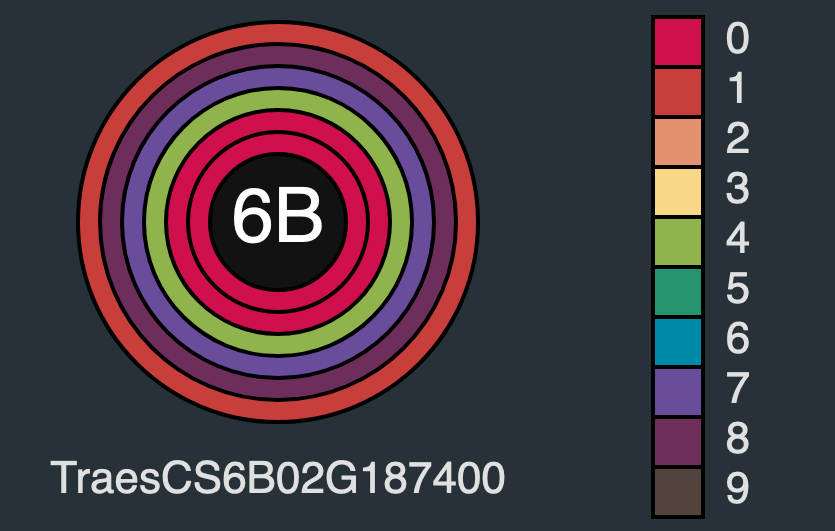
由于最近绘制基因网络图的时候,遇到了一个瓶颈。以往的都是在拟南芥中绘制的,拟南芥的基因通常拥有名称,即便没有名字,它的通用标识也非常简洁,比如AT1G04100。但是最近在涉足小麦的时候,小麦的基因一般没有公认的名称,并且他的通用标识却非常长,比如TraesCS1A02G084600,长的恶心之处就在于为了在画布中摆放这么长一串,如何在一张静止的画布中读出这个基因的标识就很困难。但是事实上在一个只有小麦基因里面这个通用标识的有效信息只有 1A 和 084600 ,但是重新组合成一个新的字符串比如 1A084600 总觉的差一口气。如果把这个信息融入在图形之中呢,就有了一个初期的想法。像电阻一样把六个数字表示成颜色串,然后就可以通过读取颜色知道这个基因的标识了。
 自然做了这样更改后,
自然做了这样更改后,R 中大部分的高度集成的软件包是不能用了,不过也想从最基础的部分去学习一下如何可视化一个复杂的网络。于是选择一个简单方便的绘图包 p5.js 去重新构建。
首先需要考虑的方面有
- 有调控关系的基因在画布上距离近,反之距离远
- 每个类的基因在画布上的距离接近
- 基因在画布中尽可能分散
首先最简单的方法就是橡皮筋模型,由于我的基因网络中的基因是通过马尔可夫聚类获得,如果能满足条件一,那么条件二就很容易满足。所以让有调控关系的基因对存在一根橡皮筋,将两者拉在一起。在这个基础上在加上一个每个基因之间的互斥的力,让基因尽可能分散。
本来觉得这个算法就非常容易实现。但是事实上还存在着一些烦人的问题,比如算法一中所提到的常数 \( c_{rep}, c_{spring} \) 预设长度 \( l \) 和时间间隔 \( \delta \),都是需要商讨。 当然在一个变种算法 Fruchterman & Reingold 中,修改了计算 \( f_{rep}, f_{spring} \) 的方式,这样就不需要 \(c_{rep}, c_{spring} \) 了。这样只剩下 \( l\) 和 \(\delta \),理智上告诉我,这里的预设长度 \( l \) 指的是最后稳定时候的平均边长,时间间隔 \( \delta \) 决定着收敛的速度,但是过大会导致震荡,所以需要做一个随时间衰减。除此之外,我为了减少初始随机条件对结果影响,加上了每个节点随着时间衰减的随机游走。最后加上一个边界限制,就得到了第一个网络图。
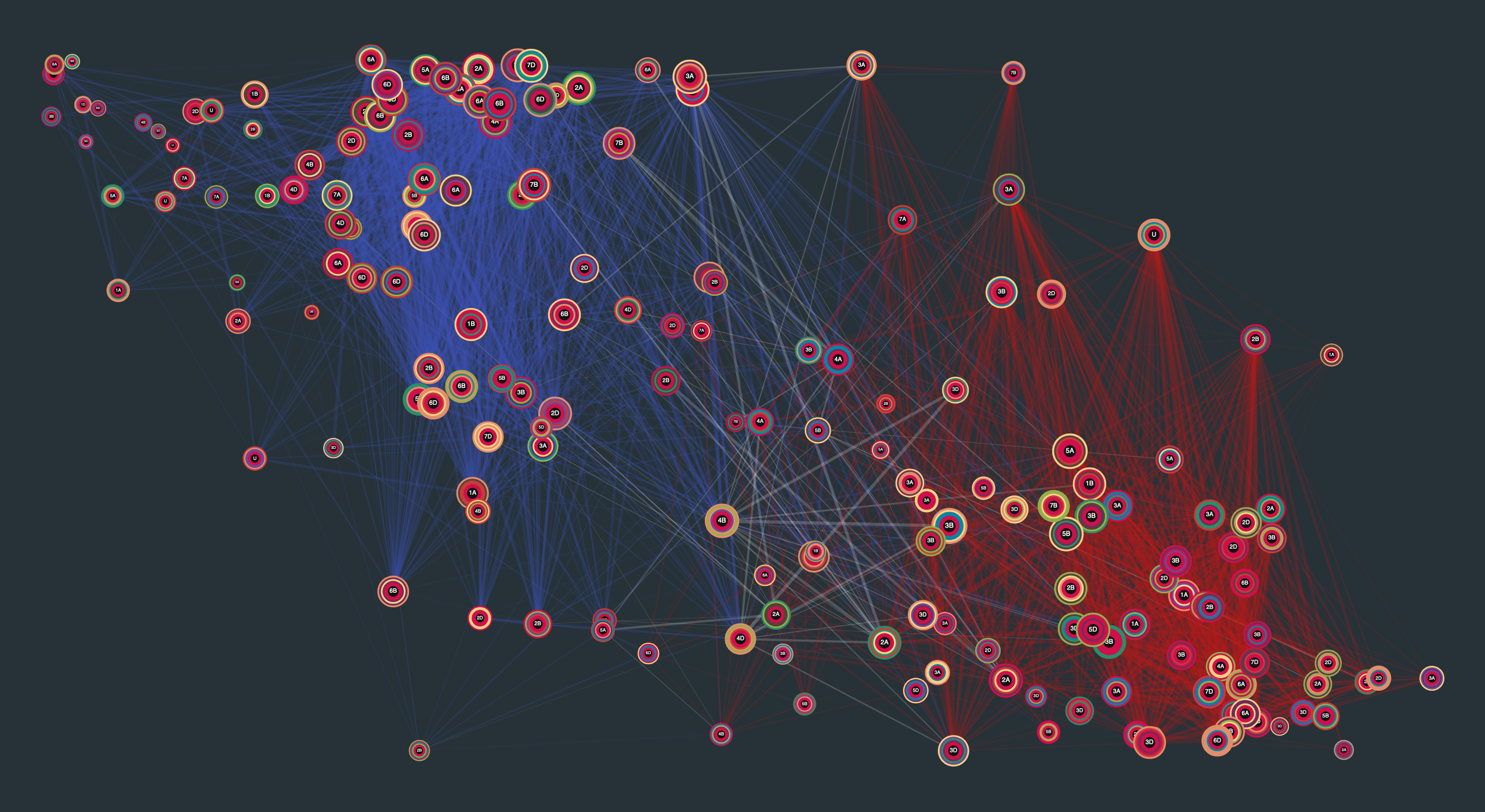 我们另外一个问题在于怎么去显示除了通用标识的信息,比如类信息。因为按照我对图形的改造,一个节点失去了最显然的特征——颜色,那么只剩下边的颜色,形状以及节点的形状了。于是我把同类相连的边涂成相同的颜色。好啦,我知道这个不好看,但是至少把这些聚成两个类这个信息表达出来了。
我们另外一个问题在于怎么去显示除了通用标识的信息,比如类信息。因为按照我对图形的改造,一个节点失去了最显然的特征——颜色,那么只剩下边的颜色,形状以及节点的形状了。于是我把同类相连的边涂成相同的颜色。好啦,我知道这个不好看,但是至少把这些聚成两个类这个信息表达出来了。
接下来需要处理的是在边界处由于强行折断导致的基因大量重叠,不过这是另外一个话题了。代码因为是自己写得爽,又乱又没有逻辑,同时是第一次写 TypeScript,就暂时不公开了,这篇文章正式结束后会贴上来吧。